Replicación
se duplica y crea así dos cadenas semejantes a la cadena original. También se entendió las diferencias de
replicación en procariontes y eucariontes, ya que en bacterias todas las
enzimas trabajan a mayor velocidad y solo existe un origen de replicación. Por
otro lado en las células eucariotas
poseen más enzimas, pero trabajan más lento, pero como presentan más
orígenes de replicación al final el proceso en eucariontes es más rápido que en
bacterias.
¿Por qué la ciencia se empeña en comprender el universo si todavia no comprende al ser humano?
viernes, 23 de marzo de 2012
Secuenciacion del DNA
Una
secuencia de ADN o secuencia genética es una sucesión de letras representando
la estructura primaria de una molécula real o hipotética de ADN o banda, con la
capacidad de transportar información.
Las
posibles letras son A, C, G, y T, que simbolizan las cuatro subunidades de
nucleótidos de una banda ADN - adenina, citosina, guanina, timina, que son
bases covalentemente ligadas a cadenas fosfóricas. En el típico caso, las
secuencias se presentan pegadas unas a las otras, sin espacios, como en la
secuencia AAAGTCTGAC, yendo de 5' a 3' de izq. a derecha.
Una
sucesión de cualquier número de nucleótidos mayor a cuatro es pasible de
llamarse una secuencia. En relación a su función biológica, que puede depender
del contexto, una secuencia puede tener sentido o antisentido, y ser tanto
codificante o no codificante. Las secuencias de ADN pueden contener "ADN
no codificante."
Las
secuencias pueden derivarse de material biológico de descarte a través del
proceso de secuenciación de ADN.
En
algunos casos especiales, las letras seguidas de A, T, C, y G se presentan en
una secuencia. Esas letras representan ambiguedad. De todas las moléculas
muestreadas, hay más de una clase de nucleótidos en esa posición. Las reglas de
la Unión Internacional de Química Pura y Aplicada (IUPAC) son las que siguen:
A
= adenina
C
= citosina
G
= guanina
T
= timina
R
= G A (purina)
Y
= T C (pirimidina)
K
= G T (keto)
M
= A C (amino)
S = G C (enlaces fuertes)
W = A T (enlaces débiles)
B
= G T C (todos y A)
D
= G A T (todos y C)
H
= A C T (todos y G)
V
= G C A (todos y T)
N
= A G C T (cualquiera)
Replicacion in Vitro del DNA (PCR)
 Es
una técnica de biología molecular desarrollada en 1986 por Kary Mullis,1 cuyo
objetivo es obtener un gran número de copias de un fragmento de ADN particular,
partiendo de un mínimo; en teoría basta partir de una única copia de ese
fragmento original, o molde.
Es
una técnica de biología molecular desarrollada en 1986 por Kary Mullis,1 cuyo
objetivo es obtener un gran número de copias de un fragmento de ADN particular,
partiendo de un mínimo; en teoría basta partir de una única copia de ese
fragmento original, o molde.
Esta
técnica sirve para amplificar un fragmento de ADN; su utilidad es que tras la
amplificación resulta mucho más fácil identificar con una muy alta
probabilidad, virus o bacterias causantes de una enfermedad, identificar
personas (cadáveres) o hacer investigación científica sobre el ADN amplificado.
Estos usos derivados de la amplificación han hecho que se convierta en una
técnica muy extendida, con el consiguiente abaratamiento del equipo necesario
para llevarla a cabo.
Pasos de la PCR.
INICIO
Este
paso consiste en llevar la reacción hasta una temperatura de 94-96 °C (ó 98 °C
si se está usando una polimerasa termoestable extrema), que se mantiene durante
1-9 minutos. Esto sólo es necesario para ADN polimerasas que requieran
activación por calor.
DESNATURALIZACIÓN
En
primer lugar, se desnaturaliza el ADN (se separan las dos hebras de las cuales
está constituido). Este paso puede realizarse de diferentes modos, siendo el
calentamiento (94-95 °C) de la muestra la forma más habitual. La temperatura a
la cual se decide realizar la desnaturalización depende, por ejemplo, de la
proporción de G+C que tenga la hebra, como también del largo de la misma. Otros
métodos, raramente empleados en la técnica de la PCR, serían la adición de
sales o agentes químicos capaces de realizar la desnaturalización.
ALINEAMIENTO
O UNIÓN DEL CEBADOR
A
continuación se producirá la hibridación del cebador, es decir, el cebador se
unirá a su secuencia complementaria en el ADN molde. Para ello es necesario
bajar la temperatura a 40-68 °C durante 20-40 segundos (según el caso),
permitiendo así el alineamiento. Los puentes de hidrógeno estables entre las
cadenas de ADN (unión ADN-ADN) sólo se forman cuando la secuencia del cebador
es muy similar a la secuencia del ADN molde. La polimerasa une el híbrido de la
cadena molde y el cebador, y empieza a sintetizar ADN. Los cebadores actuarán
como límites de la región de la molécula que va a ser amplificada.
EXTENSIÓN
O ELONGACIÓN DE LA CADENA
Actúa
la ADN polimerasa, tomando el ADN molde para sintetizar la cadena
complementaria y partiendo del cebador como soporte inicial necesario para la
síntesis de nuevo ADN. La polimerasa sintetiza una nueva hebra de ADN
complementaria a la hebra molde añadiendo los dNTP complementarios en dirección
5'→ 3', uniendo el grupo 5'-fosfato de los dNTP con el grupo 3'-hidroxilo del
final de la hebra de ADN creciente (la cual se extiende). La temperatura para este
paso depende de el ADN polimerasa que usemos. Para la polimerasa Taq, la
temperatura de máxima actividad está en 75-80 °C (comúnmente 72 °C). El tiempo
de extensión depende tanto de el ADN polimerasa usada como de la longitud del
fragmento de ADN que se va a amplificar. Hay una regla comúnmente usada: en su
temperatura óptima, la polimerasa de ADN polimerizará mil bases en un minuto.
ELONGACIÓN
FINAL
Etapa
única que se lleva a cabo a una temperatura de 70-74 °C durante 5-15 minutos
tras el último ciclo de PCR. Con ella se asegura que cualquier ADN de cadena
simple restante sea totalmente ampliado.
CONSERVACIÓN
Este
paso se lleva a cabo a 4-15 °C durante un tiempo indefinido para conservar la
reacción a corto plazo.
La
PCR normalmente se realiza con un volumen de reacción de 15-100 μL, en pequeños
tubos de 0.2-0.5 mL que se colocan en el termociclador.
Para
verificar que la PCR ha generado el fragmento de ADN previsto, se emplean
técnicas de electroforesis, que separan los fragmentos de ADN generados de
acuerdo a su carga, esto es, longitud, y, en menor medida y dependiendo de la
matriz empleada, a su tamaño: típicamente se emplean la electroforesis en gel
de agarosa, para fragmentos grandes; en acrilamida, para los más pequeños; y,
de forma más rápida y aplicable a la PCR asociada a marcaje fluorescente, la
electroforesis capilar.3 El/los tamaño/s de los productos de la PCR vienen
determinados por un marcador de peso molecular de ADN, el cual contiene
fragmentos de ADN de tamaño conocido, y que se corre en el gel junto con los
productos de PCR.
Control Genetico de la Replicacion.
El
proceso resultante de la duplicación de ADN se conoce como división celular, la
cual se ha
estudiado
a nivel citológico estableciéndose ciertas pautas cubiertas por lo que se
conoce como ciclo celular.
La
mayor parte de los estudios de ciclo celular se han llevado a cabo empleando S.
cerevisiae, la levadura del pan y la cerveza también conocida como la levadura
de gemación por su característico estilo de división. En el caso de este
organismo hay que añadir la ventaja de que actualmente se conocen los
aproximadamente 16 millones de pares de nucleótidos que constituyen la
secuencia completa de su genoma.
Los
fundamentos de nuestro conocimiento actual del ciclo celular en levaduras
vienen de la búsqueda sistemática de mutaciones en genes que codifican para
componentes de la maquinaria del ciclo.
Estos
estudios han permitido identificar una familia de proteínas quinasas
dependientes de ciclina, -Cyclin Dependent Kinases, Cdk- que incluyen a Cdc28
de S. cerevisiae, y a su homólogo en S. pombe cdc2, y que juegan un papel central
en los procesos claves del ciclo celular: la entrada en ciclo, la síntesis de
ADN y la regulación de la mitosis. Estas quinasas son las subunidades
catalíticas de un complejo que incluye también na subunidad reguladora,
denominada ciclina, necesaria para la función del complejo al determinar la
localización o la especificidad de sustrato de la quinasa.
En
los últimos años se han encontrado también homólogos a estas quinasas en todos
los eucariotas en los que se han buscado, y que incluyen desde el gusano
nematodo Caenorhabditis elegans, a la mosca Drosophila melanogaster , a
mamíferos como el ratón y el hombre y plantas como Arabidopsis thaliana,
indicando que el sistema de control del ciclo celular es general en todos los
eucariotas y validando a las levaduras como modelos de estudio.
viernes, 16 de marzo de 2012
Replicacion del Genoma Eucariotico.
Es similar a la de los procariontes, es decir, semiconservativa y bidireccional. Existe una hebra conductora y una hebra retrasada con fragmentos de Okazaki. Se inicia en ORI (puede haber unas 100 a la vez), entre las diferencia, se comienza con las polimerasas, son más complejas, y además, la polimerasa de la hebra continua es diferente a la de la hebra discontinua. De la hebra continua se encarga la polimerasa Delta y de la discontinua, la Alfa.
Las helicasa difieren en estructura, las primasas se encuentran adosadas a la ADN-pol Alfa.
El resto del proceso es muy parecido.
En el final de la hebra antigua que da la base a la hebra discontinua, queda una zona denominada telómero, que no tiene como obtener su dupla en la hebra discontinua, debido a que no es posible insertar un nuevo ARN cebador, ni mucho menos un fragmento de Okazaki. Al no tener su par, son eliminados automáticamente, perdiendose. Existen enzimas, las telomerasas, que repiten esta secuencia varias veces, cosa que no se pierda información esencial del genoma. Las enzimas encargadas de cortar el telómero, son las topoisomerasas 4.
El ARN cebador es retirado por el complejo de reparación de la célula.
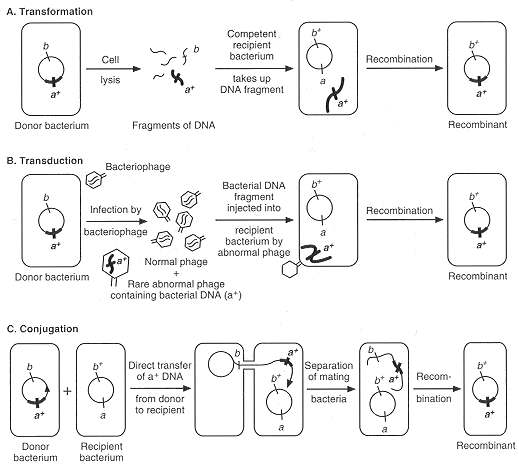
Replicacion del Genoma Procariotico.
Ocurre en tres etapas:
1ª etapa: desenrrollamiento y apertura de la doble hélice.en el punto ori.
En el punto de origen u ORI, que es un lugar del cromosoma con gran contenido de A y T, la doble hélice se abre, mediante la DNA helicasa y las proteínas desestabilizadoras de la hélice o proteínas de unión a DNA de una sola cadena, vuelven recta la cromatina y la mantienen abierta. La DNA polimerasa sintetiza las cadenas complementarias a cada una de las cadenas primitivas. Forma dos copias activas de ADN, una es continua, o sea, basta con agregar los nucleotidos correspondientes porque la hebra antigua tiene 3', por lo que se crea una 5´. En la otra hebra, se produce un proceso discontinuo, debido a que la hebra quedo con un final 5', debiendo partir con un 3', y la célula es incapaz de seguir la cadena con este final, para que se inicie la copia del DNA hace falta un corto RNA específico (10 pares de bases), denominado RNA cebador, que hace que empiece a actuar la DNA polimerasa. El RNA cebador es generado por la RNA primasa (sintetizadora de RNA). Esta enzima se une directamente a la DNA helicasa, formando un complejo llamada primosoma, que se va desplazando con la cadena en formación. Conforme van existiendo fragmentos de cadena abiertos de suficiente longitud, se va sintetizando la cadena discontinua formando pequeños fragmentos, denominados Fragmentos de Okazaki, cada uno de unos 1000 nucleótidos. . Hace falta un RNA cebador por cada fragmento de Okazaki. La RNA primasa, va sintezando a intervalos los RNA cebadores que van siendo incorporados a la copia como si fueran ADN, entre los fragmentos de Okazaki, hasta que se alcanza el RNA cebador del fragmento de Okazaki ya terminado. . La cadena con ARN cebador, es denominada cadena retrasada.
Las enzimas ligasas son las encargadas, despues, de ir arreglando los nicks cuando se sustituye el ARN cebador.
jueves, 15 de marzo de 2012
Replicacion del DNA
Es el
mecanismo que permite al DNA duplicarse (es decir, sintetizar una
copia idéntica). De esta manera de una molécula de DNA única, se obtienen dos
más "clones" de la primera.
Gracias a la complementariedad entre
las bases que forman la secuencia de cada una de las cadenas,
el DNA tiene la importante propiedad de reproducirse idénticamente,
lo que permite que la información genética se transmita de una célula madre
a las células hijas y es la base de la herencia del material
genético.
Replicación
de DNA. La doble hélice es desenrollada y cada hebra hace de plantilla para la
síntesis de la nueva cadena. La DNA polimerasa añade los nucleótidos
complementarios a los de la cadena original.
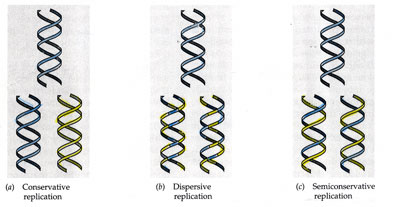
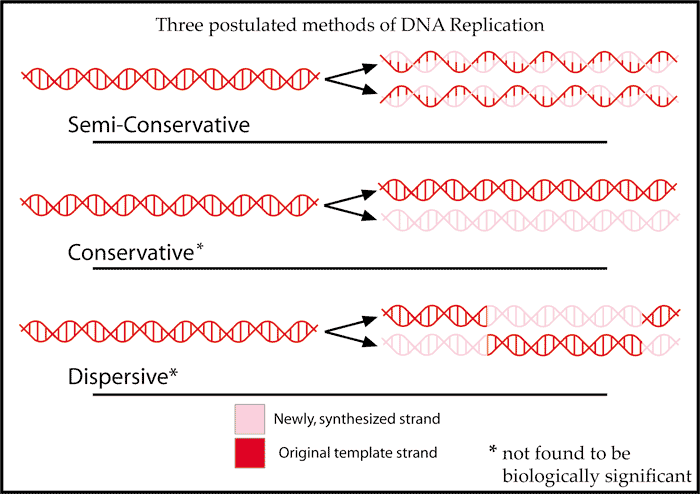
Modelos de Replicacion.
En cada
una de las moléculas hijas se conserva una de las cadenas originales, y por eso
se dice que la replicación del DNA es semi-conservadora.
- Dispersora, o dispersante. Las cadenas hijas constan de fragmentos de la cadena antigua y fragmentos de la nueva.
- Semiconservadora (modelo correcto). En cada una de las moléculas hijas se conserva una de las cadenas
Tres
posibles modelos de replicación. a) Conservadora, b) Dispersora, c)
Semiconservadora (mecanismo real)
Secuencialidad.
LA
REPLICACIÓN AVANZA EN FORMA DE HORQUILLA.
Debido a
que en la célula ambas cadenas de la doble hélice de DNA se
duplican al mismo tiempo, éstas deben separarse para que cada una de ellas
sirva de molde para la síntesis de una nueva cadena. Por eso, la replicación
avanza con una estructura en forma de horquilla.
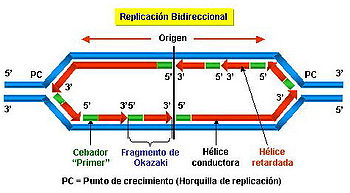
Bidireccionalidad.
El
movimiento de la horquilla es bidireccional en la mayoría de los
casos, es decir, a partir de un punto se sintetizan las dos cadenas en ambos
sentidos, que avanza en dirección a la región de DNA no duplicado dejando atrás
los dos moldes de DNA de cadena simple donde se está produciendo la
replicación.
En la
mayoría de los casos la replicación es bidireccional. No obstante, la
replicación se puede considerar, de forma general, bidireccional.
Semidescontinuidad.
La
replicación siempre se produce en sentido 5' → 3', siendo el extremo 3'-OH
libre el punto a partir del cual se produce la elongación del DNA. Esto
plantea un problema, y es que las cadenas tienen que crecer simultáneamente a
pesar de que son antiparalelas, es decir, que cada cadena tiene el extremo 5'
enfrentado con el extremo 3' de la otra cadena.
Este
problema lo resolvieron los científicos japoneses Reiji
Okazaki y Tsuneko Okazaki en la década de 1960, al descubrir que
una de las nuevas cadenas de ADN se sintetiza en forma de trozos cortos que, en
su honor, se denominan fragmentos de Okazaki.
Su
longitud suele variar entre 1000 y 2000 nucleótidos en
las bacterias y entre 100 y 400 nucleótidos en eucariontes.
DNA Polimerasa.
La DNA polimerasa es la enzima que
cataliza la síntesis de la nueva cadena de DNA a partir
de desoxirribonucleótidos y de la molécula de DNA plantilla
o molde que es la que será replicada. La enzima copia la cadena de nucleótidos
de forma complementaria (A por T, C por G) para dar a
cada célula hija una copia del DNA durante la replicación.
En cada
horquilla de replicación, la DNA polimerasa y otras enzimas sintetizan dos
nuevas cadenas de DNA que son complementarias respecto a las 2 cadenas
originales.
Su
longitud suele variar entre 1000 y 2000 nucleótidos en
las bacterias y entre 100 y 400 nucleótidos en eucariontes.
Funcion Correctora.
Además
de participar en la elongación, desempeñan una función correctora y reparadora
gracias a su actividad exonucleasa 3', que les confiere la capacidad
de degradar el DNA partiendo de un extremo de éste. Es importante que existan
estos mecanismos de corrección ya que de lo contrario los errores producidos
durante la copia del DNA darían lugar a mutaciones.
Función
correctora exonucleasa 3' → 5' de las DNA polimerasas.
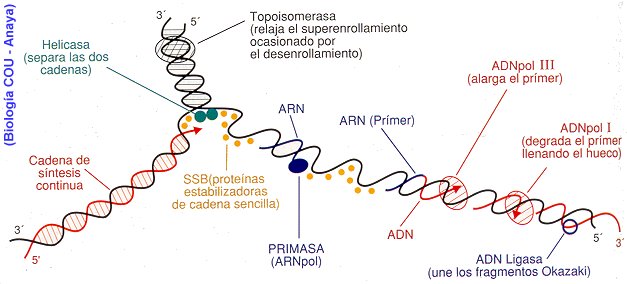
PROCESO GENERAL.
- La helicasa rompe los puentes de hidrógeno de la doble hélice permitiendo el avance de la horquilla de replicación.
- La topoisomerasa impide que el DNA se enrede debido al super-enrollamiento producido por la separación de la doble hélice.
- Las proteínas SSB se unen la hebra discontinua de DNA, impidiendo que ésta se una consigo misma.
- La DNA polimerasa sintetiza la cadena complementaria de forma continua en la hebra adelantada y de forma discontinua en la hebra rezagada.
- La RNA primasa sintetiza el cebador de RNA necesario para la síntesis de la cadena complementaria a la cadena rezagada.
- La DNA ligasa une los fragmentos de Okazaki.
INICIACIÓN.
Para que
pueda formarse la horquilla de replicación es necesario que las dos
cadenas se separen para sintetizar el cebador y el DNA de
la cadena de nueva síntesis. Para ello el DNA debe desenrollarse y el punto de
partida viene determinado por una secuencia específica
de nucleótidos conocida como origen de replicación. (Adenina y
Timina).
Proteína iniciadora: desnaturalización del
DNA y reclutamiento de proteínas. El origen de
replicación aparece en naranja.
ELONGACION.
En el
siguiente paso, la holoenzima DNA Pol III cataliza la síntesis de las
nuevas cadenas añadiendo nucleótidos sobre el molde. Esta síntesis se
da bidireccionalmente desde cada origen, con dos horquillas de
replicación que avanzan en sentido opuesto. Cuando el avance de dos
horquillas adyacentes las lleva a encontrarse, es decir, cuando dos burbujas se
tocan, se fusionan, y cuando todas se han fusionado todo el cromosoma ha
quedado replicado.
Enzimas que
participan en la replicación: helicasa, proteínas SSB,
topoisomerasa, RNA primasa, Holoenzima DNA Pol III.
Puesto
que la holoenzima ADN Pol III necesita de un extremo 3'-OH libre, es necesario
que una RNA primasa catalice la formación de un fragmento corto
específico de RNA llamado cebador, que determinará el punto por
donde la DNA polimerasa comienza a añadir nucleótidos.
Pero
debido a la unidireccionalidad de la actividad polimerasa de la DNA
Pol III, que sólo es capaz de sintetizar en sentido 5´ → 3', la replicación sólo
puede ser continua en la hebra adelantada; en la hebra rezagada es discontinua,
dando lugar a los fragmentos de Okazaki.
Hebra rezagada: síntesis
de cebadores, unión de fragmentos de Okazaki y eliminación de
los cebadores.
TERMINACIÓN.
El final de la replicación
se produce cuando la DNA polimerasa III se encuentra con una secuencia de
terminación (secuencia ter). Se produce entonces el desacople de todo el
replisoma y la finalización de la replicación.
martes, 6 de marzo de 2012
EXPERIMENTO DE MESELSON - STAHL
El experimento de Meselson--Stahl fue un
experimento realizado en 1957 por Matthew Meselson y Franklin Stahl en
el que se demostró que la replicación de ADN era semiconservadora.
Una replicación semiconservadora es aquella en que la cadena de dos filamentos
en hélice del ADN se replica de forma tal que cada una de las dos cadenas de ADN formadas consisten en un filamento
proveniente de la hélice original y un filamento nuevo sintetizado.
El experimento permitió confirmar las teorías de James Watson y
de Francis Crick sobre
el método de replicación del ADN
Para investigar la forma de
replicación del ADN, Meselsohn y Stahl idearon una forma muy ingeniosa que se
basa en dos premisas fundamentales, por una parte está el hecho de que el nitrógeno es uno de los principales elementos
del ADN, y por la otra el nitrógeno posee dos isótopos que pueden ser
distinguidos mediante técnicas de laboratorio.
De esta forma Meselsohn y Stahl mediante un inteligente
planteo del experimento, en el que utilizaron bacterias y observaron cómo
replicaban su ADN, pudieron obtener información que les permitió identificar el
mecanismo de replicación del ADN y descartar ciertas teorías alternativas.
Se
realizó un cultivo de E. coli durante varias generaciones en un medio con 15N.
Al extraer ADN de estas células y centrifugarlo en un gradiente de cloruro de
cesio, las moléculas de ADN se situaban en el punto donde su densidad igualaba
la del medio. El ADN de estas células tenía una mayor densidad que el de otras
cultivadas con 14N. Posteriormente, las células de E. coli que sólo contenían
15N en su ADN se colocaron en un medio con 14N y se les permitió que se
replicaran una sola vez. Se extrajo el ADN de estas células y se comparó con el
preparado con 14N y con el preparado con 15N, encontrando que su densidad era
prácticamente el promedio. Dado que una replicación conservadora hubiera
producido iguales cantidades de ADN de mayor y menor densidad (pero sin
producir ADN de una densidad intermedia), se descartó que la replicación
siguiera el mecanismo conservador. Sin embargo, este resultado era consistente
tanto con una replicación semiconservadora como con una de tipo dispersante.
Una replicación semiconservadora hubiera dado por resultado una doble hélice de
ADN donde un filamento tiene 15N mientras que el otro filamento tiene 14N,
mientras que una replicación dispersante hubiera dado por resultado una doble
hélice de ADN con ambos filamentos conteniendo mezclas de 15N y 14N; en ambos
casos la densidad del ADN tendría un valor promedio entre las densidades de los
ADN de 15N y 14N.
Se
extrajo ADN de células que habían crecido por varias generaciones en un medio
con 15N, y a las que se les había permitido que realizaran dos replicaciones en
un medio con 14N. Al analizar estas células se encontró que contenían
cantidades iguales de ADN con dos densidades distintas, una igual a la obtenida
tras una sola replicación en un medio con 14N, mientras que la otra
correspondía al ADN producido exclusivamente con 14N. Este resultado era
claramente inconsistente con una replicación dispersante, que hubiera dado como
producto ADN con una densidad única e inferior a la de una sola generación,
pero mayor que la de 14N, ya que el ADN original con 15N se habría repartido de
forma uniforme entre todos los filamentos de ADN. Este resultado era
consistente con una replicación semiconservadora, en cuanto a que la mitad del
ADN de segunda generación tiene un filamento original con 15N y otro con 14N,
lo que da como resultado un ADN con una densidad intermedia, mientras que la
otra mitad del ADN contiene en su totalidad 14N --un filamento sintetizado en
la primera división y otro en la segunda división. Este descubrimiento fue
sumamente importante en el desarrollo de la biología y es de gran ayuda en la
investigación y tratamiento de enfermedades.
CITA BIBLIOGRÁFICA.
Meselson, M. and Stahl, F.W. (1958). «The Replication of DNA in Escherichia coli». PNAS 44: pp. 671–82.
UNIDAD 4 REPLICACION DEL DNA
SEP SNEST DGEST
INSTITUTO TECNOLÓGICO DE CIUDAD ALTAMIRANO
UNIDAD 3
ORGANIZACIÓN DEL MATERIAL GENÉTICO
QUE PRESENTA:
GONZALEZ AGUIRRE CARLOS ALBERTO.
09930040
CARRERA
LIC. BIOLOGIA
CIUDAD ALTAMIRANO, GRO. MÉXICO. 6 MARZO 2012
OBJETIVOS.
INTRODUCCIÓN.
El proceso de replicación de ADN es el mecanismo que permite al ADN duplicarse (es decir, sintetizar una copia idéntica). De esta manera de una molécula de ADN única, se obtienen dos o más "clones" de la primera. Esta duplicación del material genético se produce de acuerdo con un mecanismo semiconservativo, lo que indica que las dos cadenas complementarias del ADN original, al separarse, sirven de molde cada una para la síntesis de una nueva cadena complementaria de la cadena molde, de forma que cada nueva doble hélice contiene una de las cadenas del ADN original. Gracias a la complementariedad entre las bases que forman la secuencia de cada una de las cadenas, el ADN tiene la importante propiedad de reproducirse idénticamente, lo que permite que la información genética se transmita de una célula madre a las células hijas y es la base de la herencia del material genético.
La
replicación del ADN produce una copia de sí mismo por medio de enzimas que
además de ser muy exactas poseen un sistema de reparación de errores. El
mecanismo de replicación es esencialmente el mismo en todas las células. Es un
proceso semiconservativo porque cada uno de los dos ADN hijos tiene una cadena
del ADN anterior
En
las células procariotas
Hay
1 lugar de origen de replicación que se muestra en la horquilla de replicación
(replication fork) y que señala el avance de la copia. La horquilla (fork)
indica que se está haciendo la separación y la replicación a la vez. El avance
es bidireccional, lo que acorta el tiempo. En el sitio en que empieza la
replicación se organizan las proteínas en un complejo llamado replisma. La
replicación del ADN en procariotas sucede a una velocidad de 500 nucleótidos
por segundo.
En
las células eucariotas
El
proceso es esencialmente el mismo pero el ADN es mucho más grADNe y linear. Hay
varios orígens de replicación y es bidireccional. El avance es más lento que en
procariotas ya que hay más proteínas asociadas al ADN que hay que soltar. La
replicación del ADN, que ocurre una sola vez en cada genración celular necesita
de muchos "ladrillos", enzimas y una gran cantidad de energía en
forma de ATP (recuerde que luego de la fase S del ciclo celular , las células
pasan a una fase G a fin de, entre otras cosas, recuperar energía para la
siguiente fase de la división celular). La replicación del ADN en el ser humano
se realiza a una velocidad de 50 nucleótidos por segundo. Los nucleótidos
tienen que ser armados y estar disponibles en el núcleo conjuntamente con la
energía para unirlos.
OBJETIVOS.
- Entenderá como se transmite la información genética entre los seres vivos y su relación con los mecanismos de herencia
- Distinguirá los mecanismos de duplicación del material genético en procariotas y eucariotas.
Conclusion
se llego a concluir que el material genetico en los organismos es de una organizacion diferente, ya que los eucariotas se empaquetan de manera diferente. Y los procariotas poseen un unico cromosoma a travez de una proteina llamada histona.
Organización Genomica Viral
Los genomas víricos pueden ser de DNA o RNA y de cadena doble o sencilla;
los genomas de RNA de doble cadena están segmentados (constituidos por distintas moléculas de RNA, cada una de ellas portadora de un determinado gen). Los genomas de RNA de cadena sencilla pueden estar segmentados
Los genomas de DNA de doble cadena pueden ser lineales o circulares.
Genes solapados: una misma secuencia puede codificar distintas proteinas funcionales dependiendo del marco de lectura escogido.
Los retrovirus poseeen un enzima denominado transcriptasa reversa capaz de sintetizar una copia de DNA a partir de una molécula de RNA.
Los virus sintetizan enormes poliproteinas que posteriormente son escindidas enzimáticamente para dar lugar a varias proteinas funcionales
DNA Mitocondrial
Es el material genético de las mitocondrias, los orgánulos que generan energía para la célula. El ADN mitocondrial se reproduce por sí mismo semi-autónomamente cuando la célula eucariota se divide. El ADN mitocondrial fue descubierto por Margit M. K. Nass y Sylvan Nass utilizando microscopia electrónica y un marcador sensitivo al ADN mitocondrial. Evolutivamente el ADN mitocondrial desciende de genomas circulares pertenecientes a bacterias, que fueron englobadas por un antiguo ancestro de las células eucarióticas.
 El ADN mitocondrial está en replicación constante, independientementemente del ciclo y del tipo celular. Se piensa que tiene lugar de forma asíncrona, es decir, que tiene lugar en las dos cadenas en tiempos diferentes y con dos orígenes distintos hacia direcciones contrarias. El comienzo tendría lugar en el origen de la cadena pesada, situado en el bucle D, y replicaría ésta tomando como molde la cadena ligera. Cuando se alcanza el segundo origen, situado a dos tercios de distancia del primero, comienza la segunda ronda de replicación en sentido opuesto. Se ha propuesto un nuevo sistema de replicación que coexistiría con el primero. Sería bidireccional y comportaría una coordinación entre hebras directas y retrasadas. En la replicación en mamíferos estarían involucradas la polimerasa y la helicasa twinkle.
El ADN mitocondrial está en replicación constante, independientementemente del ciclo y del tipo celular. Se piensa que tiene lugar de forma asíncrona, es decir, que tiene lugar en las dos cadenas en tiempos diferentes y con dos orígenes distintos hacia direcciones contrarias. El comienzo tendría lugar en el origen de la cadena pesada, situado en el bucle D, y replicaría ésta tomando como molde la cadena ligera. Cuando se alcanza el segundo origen, situado a dos tercios de distancia del primero, comienza la segunda ronda de replicación en sentido opuesto. Se ha propuesto un nuevo sistema de replicación que coexistiría con el primero. Sería bidireccional y comportaría una coordinación entre hebras directas y retrasadas. En la replicación en mamíferos estarían involucradas la polimerasa y la helicasa twinkle.
El ADN mitocondrial está sometido a un importante estrés por su proximidad con los centros de producción de radicales libres de oxígeno, de forma que disponen de una varia y compleja maquinaria de reparación, lo cual incluye diversas formas de recombinación, tanto homóloga como inhomóloga
 El ADN mitocondrial está en replicación constante, independientementemente del ciclo y del tipo celular. Se piensa que tiene lugar de forma asíncrona, es decir, que tiene lugar en las dos cadenas en tiempos diferentes y con dos orígenes distintos hacia direcciones contrarias. El comienzo tendría lugar en el origen de la cadena pesada, situado en el bucle D, y replicaría ésta tomando como molde la cadena ligera. Cuando se alcanza el segundo origen, situado a dos tercios de distancia del primero, comienza la segunda ronda de replicación en sentido opuesto. Se ha propuesto un nuevo sistema de replicación que coexistiría con el primero. Sería bidireccional y comportaría una coordinación entre hebras directas y retrasadas. En la replicación en mamíferos estarían involucradas la polimerasa y la helicasa twinkle.
El ADN mitocondrial está en replicación constante, independientementemente del ciclo y del tipo celular. Se piensa que tiene lugar de forma asíncrona, es decir, que tiene lugar en las dos cadenas en tiempos diferentes y con dos orígenes distintos hacia direcciones contrarias. El comienzo tendría lugar en el origen de la cadena pesada, situado en el bucle D, y replicaría ésta tomando como molde la cadena ligera. Cuando se alcanza el segundo origen, situado a dos tercios de distancia del primero, comienza la segunda ronda de replicación en sentido opuesto. Se ha propuesto un nuevo sistema de replicación que coexistiría con el primero. Sería bidireccional y comportaría una coordinación entre hebras directas y retrasadas. En la replicación en mamíferos estarían involucradas la polimerasa y la helicasa twinkle.
El ADN mitocondrial está sometido a un importante estrés por su proximidad con los centros de producción de radicales libres de oxígeno, de forma que disponen de una varia y compleja maquinaria de reparación, lo cual incluye diversas formas de recombinación, tanto homóloga como inhomóloga
jueves, 1 de marzo de 2012
Complejidad del Genoma
Las investigaciones llevadas a cabo hasta ahora sugieren que la complejidad del genoma humano no radica ya en el número de genes, sino en cómo parte de estos genes se usan para construir diferentes productos en un proceso que es llamado ayuste alternativo. Otra importante razón de esta complejidad radica en el hecho de que existan miles de modificaciones químicas para fabricar proteínas así como del repertorio de mecanismos que regulan este proceso.
Solenoide
La cromatina en el núcleo tiene un grosor de 300Å. La fibra de cromatina de 100Å se empaqueta formando una fibra de cromatina de 300Å. El enrollamiento que sufre el conjunto de nucleosomas recibe el nombre de solenoide.
Los solenoides se enrollan formando la cromatina del núcleo interfásico de la célula eucariota. Cuando la célula entra en división, el ADN se compacta más, formando los cromosomas.
CROMOSOMAS
El cromosoma contiene el ácido nucleico (ADN), que se divide en pequeñas unidades llamadas genes. Éstos determinan las características hereditarias de la célula u organismo. Las células de los individuos de una especie determinada suelen tener un número fijo de cromosomas, que en las plantas y animales superiores se presentan por pares.
El ser humano tiene 23 pares de cromosomas. En estos organismos, las células reproductoras tienen por lo general sólo la mitad de los cromosomas presentes en las corporales o somáticas. Durante la fecundación, el espermatozoide y el óvulo (células reproductoras o gametos) se unen y reconstruyen en el nuevo organismo la disposición por pares de los cromosomas; la mitad de estos cromosomas procede de un parental, y la otra mitad del otro.
Los cromosomas se duplican al comienzo de la división celular y, una vez completada, recuperan el estado original.
Debido a esta duplicación, que aparece con la forma de una X, se llama cromosoma a esta cadena duplicada de ADN, que aparece constituida por dos partes idénticas, denominadas cromátidas, que se unen a través de una zona de menor densidad, y un centro llamado centrómero. Los elementos separados por el centrómero hacia arriba y hacia abajo de cada cromátida reciben el nombre de brazos (corresponden a la mitad de una cromátida).
Es en la metafase cuando las cromátidas están duplicadas (cromátidas hermanas) y unidas a nivel del centrómero. Luego, durante la anafase sólo presenta un juego de cromátidas.
El tamaño de los cromosomas puede oscilar entre los 0,2 y 5 µm (micrómetros) de longitud con un diámetro entre 0,2 y 2 µm. La longitud normal de los cromosomas de los mamíferos varía entre los 4 a 6 µm.
Un micrómetro equivale a la milésima parte de un milímetro. (Un milímetro dividido en mil partes)
Normalmente existen 46 cromosomas en cada célula humana.
Estas instrucciones son llamadas "genes". Por lo tanto, los cromosomas son paquetes de genes los cuales dirigen el desarrollo del cuerpo.
HISTONAS
Las proteínas celulares más frecuentes son las proteínas histonas, siendo que cada célula eucariótica presenta varios cientos de millones de moléculas de histonas, mientras que las demás proteínas no alcanzan unos cientos (como mucho, a miles). Son proteínas de masa molecular baja, aproximadamente 11-12 Kd y exhiben un alto contenido, cerca de 20%, de lisina y arginina (aminoácidos básicos). Con las cargas positivas de las cadenas laterales de estos restos, las histonas (que son extremadamente básicas) se unen a los grupos fosfato del ADN (cargados negativamente); para ello no es relevante la secuencia de bases dentro del ADN. A menudo, las histonas son modificadas por metilaciones, acetilaciones, fosforilaciones o ADP-ribosilaciones.
–Metilaciones: Determinan cambios permanentes en la cromatina. Están destinadas al mantenimiento de un tipo determinado de expresión génica (Enzimas encargadas: HMTasas).
–Metilaciones: Determinan cambios permanentes en la cromatina. Están destinadas al mantenimiento de un tipo determinado de expresión génica (Enzimas encargadas: HMTasas).
–Acetilaciones: en las colas de las histonas a nivel de lisina y arginina: modifican la cromatina determinando que pueda transcribir (Enzimas encargadas: HAT).
–Desacetilaciones: determinan la compactación de la cromatina, silencia la actividad transcripcional (Enzimas encargadas: HDAC)
En los seres humanos hay cinco tipos principales: la histona H1 y las histonas H2A, H2B, H3 y H4. Estas últimas se denominan también histonas nucleosomales y forman un octámero con dos histonas de cada; alrededor de este núcleo se enrolla dos veces un hilo de ADN. Este complejo ADN-histona recibe el nombre de nucleosoma y constituye el componente primario del cromosoma.
El ADN gira unos 147 pares de bases alrededor del núcleo de la histona y a continuación se desplaza unos 20-70 bp en un giro hacia la izquierda hasta alcanzar el siguiente nucleosoma. La pieza intermedia, también denominada ADN de conexión está “desnuda”, es decir, no está equipada con histonas. La histonas H1 se coloca como pieza de cierre en cada nucleosoma y al mismo tiempo toma contacto con las agrupaciones vecinas. De esto modo, las proteínas H1 van “grapando” los nucleosomas para formar un hilo denso: la fibra de cromatina.
DNA Lineal y Empaquetamiento.
Cada molécula de ADN se empaqueta en un cromosoma el total de la información genética contenida en los cromosomas de un organismo, constituye su genoma.
Los cromosomas de una célula, en mitosis o fase M, se encuentran muy condensados y son transcripcionalmente activos.
En la interfase, están mucho menos condensados y son activos dirigiendo continuamente la síntesis de RNA.
 Cada molécula de ADN que forma un cromosoma ha de contener un centromero, dos telómeros y varios orígenes de replicación.
Cada molécula de ADN que forma un cromosoma ha de contener un centromero, dos telómeros y varios orígenes de replicación.
Para que una molécula de ADN forma un cromisoma funcional ha de ser capaz de dirigir la sintesis de ARN y de propagarse, transmitiendose eficasmente de una generación a otra.
Necesitan un origen de replicación, centrómero que une la molécula de ADN que lo contiene al huso mitótico durante la divición celular telómero eciste uno de ellos encada extremo del cromosoma lineal.
ORGANISMOS EUCARIOTICOS.
 Son aquellos en cuyas células puede diferenciarse un núcleo que contiene el material genético separado de un citoplasma en el que se encuentran diferentes orgánulos celulares.
Son aquellos en cuyas células puede diferenciarse un núcleo que contiene el material genético separado de un citoplasma en el que se encuentran diferentes orgánulos celulares.
Los organismos eucariotas incluyen algas, protozoos, hongos, plantas superiores, y animales. Este grupo de organismos posee un aparato mitótico, que son estructuras celulares que participan de un tipo de división nuclear denominada mitosis; tal como imnúmeras organelas responsables de funciones específicas, incluyendo mitocondrias, retículo endoplasmático y cloroplastos.

Los cromosomas se encuentran en el núcleo celular separados del resto de la célula por la membrana nuclear. Un cromosoma tiene tres partes fundamentales: centrómero, telómero y los brazos.
El centrómero (constricción cromosómica primaria) es la estructura a la que se une el huso acromático. La región centromérica aparece normalmente como un y su posición define la relación entre las longitudes de los dos brazos centroméricos que es una característica muy útil. Según la posición del centrómero los cromosomas se clasifican en:
- Telocéntricos con el centrómero en un extremo.
- Acrocéntricos con el centrómero alejado del centro.
- Metacéntricos con el cromosoma en el centro.
El número de nucleolos difiere de un organismo a otro, variando entre uno y muchos. Los nucleolos contienen ARN ribosómico, un componente muy importante de los ribosomas. Los nucleolos se encuentran situados en las constricciones secundarias de los cromosomas llamadas organizadores nucleolares, que ocupan lugares específicos en el cromosoma.
En los centrómeros y telómeros se encuentra asociado ADN satélite que son segmentos de ADN altamente repetitivo y moderadamente repetitivo.Los cromosomas eucarióticos están la mayor parte del ciclo celular como una sola cromátida y como dos cuando se replica. La replicación del ADN es semiconservativa, esto se demostró en un experimento en el que se marcó con tritio una cromátida. Entonces se procedió a replicar esta cromátida en presencia de tritio y se obtuvo un cromosoma de dos cromátidas marcadas. Se hizo volver a replicarse, esta vez sin presencia de tritio y se obtuvieron cuatro cromátidas formando dos cromosomas. Cada cromosoma tenía una molécula marcada y la otra no con lo que en la replicación se conservaba para el nuevo cromosoma una de las cromátidas parentales.
Suscribirse a:
Entradas (Atom)