En esta unidad8 vimos que el operon lactosa en eucariotas son
varios puntos en los que se puede controlar la expresión génica que
son: Control pretranscripcional, control transcripcional y control
postranscripcional. Durante la unidad aprendimos
algunos términos nuevos, tales como son el promotor que es una
secuencia reconocida específicamente por la RNA polimerasa para iniciar la
transcripción de una unidad de transcripción, otro es el operador que es una
secuencia del promotor que es reconocida por una proteína reguladora. Y
aprendimos que un Operón es grupo de genes estructurales cuya expresión está
regulada por los mismos elementos de control promotor y operador y genes
reguladores. El otro operón que vimos fue el del Triptofano que es un
sistema de tipo represible, ya que el aminoácido triptófano que es correpresor,
este impide la expresión de los genes necesarios para su propia síntesis cuando
hay niveles elevados de triptófano, y cuando hay ausencia de triptofano es
cuando existe transcripción.sobre la regulación de la transcripción en
organismos eucarióticos vimos que existen varias señales que modifican
dicha transcripción, que son señales hormonales y señales nutricionales.
¿Por qué la ciencia se empeña en comprender el universo si todavia no comprende al ser humano?
martes, 29 de mayo de 2012
8.2.1 Operon De Lactosa.
Un Operón es grupo de genes estructurales cuya expresión está regulada por los mismos elementos de control (promotor y operador) y genes reguladores.
Los principales elementos que constituyen un operón son los siguientes:
·
Los
genes estructurales: llevan información para polipéptidos. Se
trata de los genes cuya expresión está regulada. Los operones bacterianos
suelen contener varios genes estructurales, son poligénicos o policistrónicos.
Hay algunos operones bacterianos que tienen un solo gene estructural. Los operones
eucarióticos suelen contener un sólo gen estructural siendo monocistrónicos.
·
El
promotor (P): se trata de un elemento de control que es
una región del ADN con una secuencia que es reconocida por la ARN polimerasa
para comenzar la transcripción. Se encuentra inmediatamente antes de los genes
estructurales. Abreviadamente se le designa por la letra P.
·
El
operador (O): se trata de otro elemento de control que
es una región del ADN con una secuencia que es reconocida por la proteína
reguladora. El operador se sitúa entre la región promotora y los genes
estructurales. Abreviadamente se le designa por la letra O.
·
El
gen regulador (i): secuencia de ADN que codifica para la
proteína reguladora que reconoce la secuencia de la región del operador. El gen
regulador está cerca de los genes estructurales del operón pero no está
inmediatamente al lado. Abreviadamente se le denomina gen i.
·
Proteína
reguladora: proteína
codificada por el gen regulador. Está proteína se une a la región del operador.
·
Inductor: sustrato o compuesto cuya presencia induce
la expresión de los genes.
El Operón lactosa, que abreviadamente se denomina Operón lac, es un sistema inducible que está bajo control negativo, de manera que la proteína reguladora, producto del gen regulador i, es un represor que impide la expresión de los genes estructurales en ausencia del inductor. El inductor del sistema es la lactosa. Como veremos más adelante, eloperón lac también está bajo control positivo, ya que existe otra proteína que estimula la transcripción de los genes estructurales.
Los genes estructurales del operón lactosa son los siguientes:
·
El gen z+: codifica para la b-galactosidasa que cataliza la hidrolisis de la lactosa en
glucosa más galactosa.
·
El gen y+: codifica
para la galactósido
permeasa que transporta b-galactósidos al interior de la
célula bacteriana.
·
El gen a+: codifica para la tiogalactósido transacetilasa que cataliza la transferencia del
grupo acetil del acetil Coenzima A al 6-OH de un aceptor tiogalatósido. Este
gen no está relacionado con el metabolismo de la lactosa.
El verdadero inductor del sistema es la Alolactosa y no la lactosa de manera que la β-galactosidasa transforma la lactosa en Alolactosa. En los
estudios del operón lactosa se utiliza como inductor un análogo sintético de la
lactosa que es el Isopropil
tiogalactósido (IPTG). El IPTG no necesita ser transportado por la galactósido permeasa para entrar en la bacteria.Las cepas normales de E. coli son inducibles, de manera que en ausencia del inductor (la lactosa), la proteína represora producto del gen i se encuentra unida a la región operadora e impide la unión de la ARN-polimerasa a la región promotora y, como consecuencia, no se transcriben los genes estructurales.
Operon lactosa sin lactosa.
Sin embargo, en presencia del inductor (la lactosa), este se une a la
proteína reguladora que cambia su conformación y se suelta de la región
operadora dejando acceso libre a la ARN-polimerasa para que se una a la región
promotora y se transcriban los genes estructurales. Por consiguiente, la
presencia del inductor hace que se expresen los genes estructurales del operón,
necesarios para metabolizar la lactosa.
Literatura Citada.
http://www.ucm.es/info/genetica/grupod/Operon/Operon.htm
8.2 Regulación De La Transcripción En Organismos Procarióticos.
Unidad de Transcripción: Los genes/cistrones/ORF
que se transcriben bajo en control de un mismo promotor
Promotor: Secuencia reconocida específicamente por
la RNA polimerasa para iniciar la transcripción de una unidad de transcripción
Cistrón :
Cada una de las partes de un transcrito que darán una biomolécula funcional
(proteína o RNA).
Terminador: Conjunto de secuencias que marcan el
fin de la transcripción de una unidad de transcripción.
Operón: La unidad de transcripción (promotor,
cistrones y terminador) junto con las secuencias/genes adicinales que sirven
para regularlo
Polaridad: Es el efecto de una mutación en un gen
sobre la expresión de otros cistrones distales dentro del mismo operón
Proteína Reguladora: Aquella que controlará la
expresión de un operón. Pueden ser activadoras o represoras
Operador: Secuencia del promotor que es reconocida
por una proteína reguladora.
Efector :
Biomolécula no proteica que controla la activación o inactivación de la
proteína reguladora. Las hay inhibidoras y corepresoras.
Regulón: Conjunto de genes/operones que responden
al unísono por la acción de un regulador
Cambios en la estructura del DNA
Metilación.
La metilación provoca un cambio de estructura en el
apareamiento entre las bases nitrogenadas que puede alterar su reconocimiento
por algunas proteínas. El más conocido es el de la metilasa dam que reconoce la
secuencia GATC y metila la A.
La mayor parte de los genes cuya exprexión se ve
reprimida por la metilación son genes cuya expresión sólo se necesita durante
la replicación (único momento en el que una cadena del DNA está
transitoriamente hemimetilado), permaneciendo reprimidos el resto del ciclo
celular.
Superenrollamiento.
Para mantener una situación homeostática en la
célula en relación al número de superenrollamientos es necesario mantener con
una regulación contraria los genes “topa” que codifica la topoisomerasa I y
“gyrA” y “gyrB” que determinan las dos subunidades de la DNA-topoisomerasa II.
No se conoce el mecanismo molecular que controla esta regulación. Sí se sabe
que mutantes en las topoisomerasas disminuyen la tasa general de transcripción.
Cambios en la interacción entre el DNA y la
RNA-polimerasa.
Lo provocan aquellos cambios que, sin alterar ni la
estructura del DNA ni la de la RNA-polimerasa, sí que afectan la interacción
entre ambas. Es necesaria la comparecencia de una tercera molécula,
habitualmente una proteína aunque a veces puede ser RNA.
Cita Bibliografica.
Bibliografía:
http://biomoleculi.galeon.com/tres.htm
8.1 Niveles De Regulación De La Expresión Genética.
La
estrategia procariota pretende alcanzar las máximas tasas de proliferación
cuando el entorno se lo permita.
En
cambio, la estrategia eucariota ha de ser distinta puesto que en los organismos
pluricelulares donde el medio intercelular es relativamente constante, el
control génico está al servicio de la especialización celular.
Así,
se encuentran genes que no responden a cambios fisiológicos y otros que sufren
un fuerte control como consecuencia del desarrollo, de la organización de
células en tejidos, y de los tejidos en organismos completos.
La
genómica indica:
El
número de genes no varía mucho entre las especies: los vertebrados tienen como
mucho el doble de genes que los invertebrados;
El
número de genes no sirve para explicar la diversidad evolutiva por mutación o
duplicación génica;
La
variabilidad de los genes se debe a la duplicación de genes en vez de la
creación de genes nuevos.
La
complejidad evolutiva se correlaciona con el aumento de genes reguladores: en
las levaduras hay un gen regulador por cada 20 funcionales, pero en humanos hay
más de 3 000 reguladores para unos 30 000 genes
Es
por esto que la complejidad de los organismos emerge de una regulación de la
expresión génica cada vez más elaborada y no de cambios o mutaciones en los
genes estructurales o enzimáticos: la secuencia de las proteínas se conserva
mucho a través de distintas especies, sin cambios importantes mientras que los
cambios en el orden de los elementos del promotor o de sus elementos
reguladores provocan alteraciones drásticas.
Cita Bibliografica.
Bibliografía:
http://med.unne.edu.ar/catedras/bioquimica/expresion.htm
SEP SNEST DGEST
INSTITUTO TECNOLÓGICO
DE CIUDAD ALTAMIRANO
UNIDAD 8 REGULACIÓN
DE LA EXPRESIÓN GENÉTICA
Nombre del Alumno: González
Aguirre Carlos A.
Número de Control:
09930040
Carrera: Lic.
Biología
Ciudad Altamirano,
Gro. México. 24 de Mayo del 2012
INTRODUCCION.
Los
organismos multicelulares complejos están compuestos de diferentes tejidos
cuyas características individuales dependen de las proteínas específicas
expresadas por sus tipos celulares. La diferenciación, el desarrollo y la funcionalidad de los tejidos específicos dependen del conjunto de proteínas selectivamente expresadas
por cada célula. Estas proteínas expresadas en forma diferencial pueden funcionar como componentes estructurales de
las células, enzimas reguladoras del metabolismo, factores de
transcripción, receptores celulares,
componentes intracelulares de señalización, etc. La expresión incorrecta de
tales proteínas, su expresión en lugares equivocados, a destiempo, o la
producción en cantidades anormales de proteínas específicas o de proteínas de
función anómala subyace a toda patología celular de base genética.
Objetivo.
Integrar
los Conocimientos anteriores con los mecanismos de regulación genética para
entender a nivel molecular los procesos metabólicos.
Metodología
Para
la realización de la unidad número 8 se tomarán referencias bibliográficas de;
miércoles, 23 de mayo de 2012
Tareaa...!
Es Posible Que La Maquinaria Eucarionte De Traducción Pueda Traducir El mRNA De Una Bacteria?
Podemos decir que se pueden diferenciar en los
procesos de iniciación, ya que en las bacterias la subunidad menor se une, de
forma directa, a la región que rodea el codón de iniciación mediante puentes de
hidrogeno entre las secuencias consenso Shine-Delgarno en la región 5´.
No lo
creo.
Porque?
En el código
genético de procariotas y eucariotas es idéntico, a excepción de que en hay un aminoácido
en especificado en el codón de inicio en las eucariotas.
También podemos
decir que el RNA en procariotas puede tener un periodo de longevidad muy corto
(de minutos), mientras que en las procariotas, estas pueden tener un periodo de
longevidad que puede durar varios días.
Conclusion...!
En
esta unidad emos concluido que la síntesis de proteinas es un proceso mediante
el cual el RNA se transforma en proteínas, y para ello, este proceso se da en
los ribosomas. También hemos comprendido que existen diferencias entre los
ribosomas de procariontes y eucariontes, ya que en las bacterias los ribosomas
son de menor tamaño, y ademas estos organelos presentan un sitio E. Otro caso
muy curioso es que en el momento de la elongación en la síntesis de proteínas,
los ribosomas se mueven a través del ARN. Por ultimo también se comprendió que en el código genético existen
64 tripletes, tres de ellos de parada y el resto de todos ellos son los que
codifican los 20 aminoacidos, y por mencionar al triplete de AUG o metionina es
el aminoácido de inicio en toda
traducción.
jueves, 17 de mayo de 2012
7.4.1 Modificacion De Proteinas Postraduccion..!
PLEGAMIENTO.
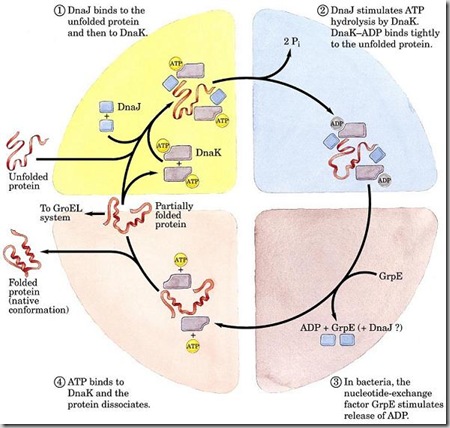 Todavía no se sabe con certeza cómo la
información de una secuencia primaria de aminoácidos guía su estructura
tridimensional. El plegamiento comienza en cuanto se sintetizan más de 30 aa
—son los que el ribosoma protege—. Por ejemplo, la ß-galactosidasa comienza a
formar los tetrámeros antes de terminar cada monómero.
Todavía no se sabe con certeza cómo la
información de una secuencia primaria de aminoácidos guía su estructura
tridimensional. El plegamiento comienza en cuanto se sintetizan más de 30 aa
—son los que el ribosoma protege—. Por ejemplo, la ß-galactosidasa comienza a
formar los tetrámeros antes de terminar cada monómero.
La importancia del plegamiento se
refleja en que algunas enfermedades tienen su base molecular en proteínas mal
plegadas:
La enfermedad de Alzheimer se origina
en la acumulación de la proteína ß-amiloide mal plegada.
La enfermedad de las vacas locas,
scrapie en ovejas o enfermedad de Creutzfeld-Jacob se debe a la acumulación de
la proteína priónica PrP plegada incorrectamente que además cataliza la
conversión de las PrP bien plegada en una PrP mal plegada.
Principios del plegamiento
La secuencia de mecanismos que
intervienen en el plegamiento de las proteínas ha sido descubierta gracias a
experimentos clásicos de desnaturalización y renaturalización de proteínas
pequeñas, la aplicación de métodos biofísicos de análisis, y la construcción de
mutantes específicos. Las etapas iniciales del plegamiento son:
1.- La formación de las estructuras
secundarias. Con ello se disminuye la entropía (ΔS), por lo que es necesario
que la entalpía (ΔH) sea muy negativa para que la energía libre final (ΔG) sea
negativa y permita que se formen.
2.- Eliminación de las interacciones de
los residuos hidrófobos con el disolvente acuoso. De esta forma se establece la
estructura terciaria.
El ejemplo mejor conocido es el de la
proteína GroEL (Hsp60 bacteriana) de E. coli con la proteína acompañante GroES
(Hsp10). El plegamiento se produce en el anillo toroidal superior.
GLUCOSILACION.
La glucosilación es la adición de uno o
más glúcidos a una proteína lo que da lugar a las glucoproteínas, que son
esenciales en los mecanismos de reconocimiento celular. La glucosilación puede
implicar la adición de unas pocas moléculas glucídicas o de grandes cadenas
ramificadas de oligosacáridos. Existe un centenar de glucosiltransferasasdistintas,
las enzimas encargadas de realizar este proceso. El mecanismo es básicamente el
mismo en todos los casos; un azúcar es transferido desde un sustrato dador
activado hasta un aceptor apropiado.
7.4 Etapas De La Sintesis De Proteinas En Organismos Eucarioticos...!
El mecanismo de eucariotas es
básicamente el mismo que el mecanismo de procariotas, solo con la mayor parte
de las diferencias acumuladas en la iniciación.
INICIACIÓN.
A continuación, el primer ARNt
iniciador se coloca en su lugar y se aparea con el codón iniciador del ARNm.
Este codón iniciador que habitualmente es (5´)-AUG-(3´), se aparea en forma
antiparalela con el anticodón del ARNt (3´)-UAG-(5´). El ARNt iniciador
entrante, que se une al codón AUG, lleva una forma modificada del aminoácido
metionina, N-formilmetionina o fMet. Esta fMet será el primer aminoácido de la
cadena polipeptídica recién sintetizada que es rápidamente removido.
El ARNt iniciador está ubicado en el
sitio P de la subunidad mayor, uno de los dos sitios de unión para las
moléculas de ARNt. Luego, se liberan estos factores de iniciación y la
subunidad ribosómica mayor se une a la subunidad menor. La energía para este
paso la suministra la hidrólisis del trifosfato de guanosina.
ELONGACION.
Una vez que el ribosoma completo se ha
ensamblado en el codón de inciación, comienza la etapa de elongación o
alargamiento. Durante esta etapa, elsitio A de un ribosoma será ocupado
transitoriamente por sucesivos aminoacil.ARNt.Los aminoacil-ARNt que ocupen el
sitio A serán aquellos cuyo anticodón sea complementario al codón que queda
expuesto en ese sitio. La entrada del aminoacil-ARNt al sitio A del ribosoma
requiere su unión previa con una proteína llamada factor de elongación, que en
su forma activa será unida al GTP. Al aparearse el ARNt con el ARNm, se dispara
la hidrólisis del GTP por parte del factor de elongación que luego se disocia,
permitiendo que el aminoacil-ARNt permanezca unido por un corto período al
ARNm.
Cuando los sitios A como P están
ocupados, una enzima, la peptidil transferasa, que es parte de la subunidad
mayor del ribosoma, cataliza la formación de un enlace peptídico entre los dos
aminoácidos, acoplando el primero (fMet) al segundo. El primer ARNt, entonces
se libera. El ribosoma se mueve un codón a lo largo de la cadena de ARNm; en
consecuencia, el segundo ARNt, al cual ahora se encuentran acoplados la fMet y
segundo aminoácido, se transfiere de la posición A a la posición P. Un tercer
aminoacil-ARNt se ubica en la ahora vacante posición A, apareado al tercer
codón del ARnm, y se repite el paso. La posición P acepta al ARNt que carga con
la cadena polipeptídica creciente; la posición A acepta al ARNt que porta el
nuevo aminoácido que será añadido a la cadena.
TERMINACION.
Cuando el mensaje llega a un codón de
terminación, ya no hay ningún ARNt que tenga su anticodón, de manera que ya no
se agrega otro aminoácido a la cadena. Se libera el polipéptido formado, la
proteína. Se separan las dos subunidades del ribosoma y el ARNm es liberado.
7.3 Etapas De La Sintesis De Proteinas En Organismos Procarioticos...!
INICIO (Traduccion)
La primera etapa comienza con la
subunidad menor sola. El IF-1 se une a la base del sitio A para forzar que el
primer fMet-tRNA entre en el sitio P.
El IF-3 se necesita para estabilizar la
subunidad 30S, que sirve para depositar el aminoacil-tRNA Met-tRNA en este caso
en el ribosoma.
Los 3 IF junto con el mRNA, el
fMet-tRNA y la subunidad 30S forman el complejo de iniciación. El tRNA
iniciador que reconoce primeramente el AUG en ocasiones GUG y raramente UUG, es
especial ya que porta una formil-Met, presenta modificaciones
postranscripcionales específicas, sólo puede usarse en iniciación, y es el
único capaz de entrar en el sitio P, sin la subunidad mayor del ribosoma.
LA ELONGACIÓN
El crecimiento de la cadena
polipeptídica en el ribosoma es un proceso cíclico ya que se repite tantas veces
como aminoácidos se incorporen.
Cada ciclo consta de 4 pasos que son:
(Ubicación)
El tRNA aminoacilado se dirige al sitio
A con el factor de elongación EF-Tu, que al igual que IF-2, lleva GTP. Cuando
el aminoacil-tRNA se aloja en el sitio A, el GTP se hidroliza y se libera.
(Corrección)
Para dejar el aa-tRNA en su sitio, se
tiene que hidrolizar el GTP. Esto es un proceso relativamente lento, que da
tiempo a verificar el apareamiento codón-anticodón. Si el apareamiento
codón-anticodón es incorrecto, el aminoacil-tRNA se rechaza y queda de nuevo
libre el sitio A para aceptar el aminoacil-tRNA correcto.
(Transpeptidación)
La cadena polipeptídica enganchada al
tRNA del sitio P se transfiere sobre el aminoácido transportado por el tRNA del
sitio A. Esta transferencia la cataliza el sitio peptidil transferasa de la
subunidad 50S. Concretamente, el rRNA 23S alojado en este sitio catalítico es
quien realiza la función catalítica fundamental, actuando como ribozima.
(Translocación)
El tRNA descargado del sitio P se
transfiere al E y el tRNA que tiene el péptido en el sitio A pasa al P. El
desplazamiento hace que el ribosoma avance 3 nt por el mRNA.
Tras la translocación, la cooperación
negativa entre E y A hace que no pueda entrar otro aa-tRNA nuevo en A hasta que
el que hay en E no ha salido. En este momento se ha completado el ciclo, con la
diferencia de que ahora la cadena polipeptídica ha crecido en un residuo y el
ribosoma está desplazado 3 nt en el mRNA.
FINAL (Terminacion)
Determina la síntesis de la proteína
cuando el sitio A del ribosoma es abordado por el codón de terminación del
ARNm (UUA, UGA o UAG, indistintamente).
Ello deja al sitio A sin el esperado aminoacil-ARNtAA, aunque pronto es ocupado
por un factor de terminación llamado eRF, que sabe reconocer a los tres codones
de terminación.
En síntesis la terminación de la cadena
polipeptídica está señalada por el ARNm mediante un codón que no especifica la
incorporación de ningún aminoácido . Ese codón de terminación puede ser UUA,
UGA o UAG, y sobre él no se une ningún ARNt.
LITERATURA CITADA:
http://payala.mayo.uson.mx/QOnline/sintesisproteinas.html
7.2.3 Procariotico..! & 7.2.4 Eucariotica..!
LAS BACTERIAS
Son células sin núcleo, la zona de la
célula, donde está el ADN y ARN no está limitado por membrana. Ej. Bacteria.
Actualmente están divididas en dos
grupos:
• Eubacterias, que poseen paredes
celulares formadas por peptidoglicano o por mureína. Incluye a la mayoría de
las bacterias y también a las cianobacterias.
• Arqueobacterias, que utilizan otras
sustancias para constituir sus paredes celulares. Son todas aquellas
características que habitan en condiciones extremas como manantiales sulfurosos
calientes o aguas de salinidad muy elevada. La celula procariota, también
procarionte, organismo vivo cuyo núcleo celular no está envuelto por una
membrana, en contraposición con los organismos eucariotas, que presentan un
núcleo verdadero o rodeado de membrana nuclear.
7.2.4 Eucariotica...!
La estructura en eucariotas es básicamente la misma
estructura que el de las procariotas, solo presentan algunas diferencias:
·
El ribosoma y sus subunidades son más
grandes (40S + 60 S → 80S).
·
Los rRNA son mayores y hay más
proteínas por subunidad ribosómica.
·
La subunidad mayor contiene los rRNA
28S y 5S, pero además una 5,8S adicional que no existe en los procariotas.
·
El ribosoma no tiene sitio E.
LITERATURA CITADA:
http://aulavirtual.usal.es/aulavirtual/demos/biologia/modulos/curso/uni_01/u1c3s2.htm
7.2.3 Estructura Ribosomal..!
El
ribosoma consta de dos partes, la subunidad mayor y una menor, estas salen del
núcleo por separado.
El
ribosoma procarionta tiene un coeficiente de sedimentación de 70s y está
formado por dos subunidades de 50s y 30s. Se puede encontrar en el citoplasma
donde recibe el nombre de polisoma o polirribosa.
El
ribosoma eucariota tiene un coeficiente de sedimentación de 80s, uno de 60s y
otra de 40s Este se puede encontrar unido al retículo endoplasmático rugoso.
La
unión de ambas subunidades se realiza en presencia de una concentración 0.001 M
de iones Mg, si esta concentración disminuye se produciría la separación de las
dos subunidades, es por lo tanto un proceso reversible.
Los
ribosomas son responsables del aspecto granuloso del citoplasma de las células.
Es el orgánulo más abundante, hay varios millones por célula.
LITERATURA CITADA:
http://www.mitecnologico.com/ibq/Main/EstructuraYFuncionDeLosRibosomas
7.2.1 Tipos De RNA...!
El
ácido ribonucleico (ARN o RNA,) es un ácido nucléico formado por una cadena
deribonucleótidos. Está presente tanto en las células procariotas como en las
eucariotas, y es el único material genético de ciertos virus (virus ARN). El
ARN celular es lineal y de hebra sencilla, pero en el genoma de algunos virus
es de doble hebra.
Hay
4 tipos de ARN, cada uno codificado por su propio gene:
- ARNm - ARN Mensajero: Codifica la secuencia de aminoácido de un polipéptido.
- ARNt - ARN de Transferencia: Lleva los aminoácidos a los ribosomas durante la traducción.ARNr -
- ARN Ribosomal: Con proteínas ribosomales y los ribosomas actúan con el ARNm.
- ARN np- ARN nuclear pequeño: Con proteínas, forma complejos que son usados en el proceso de ARN en las células eucarióticas (no se encuentra en las células procarióticas).
LITERATURA CITADA:
Vázquez, F., Vaucheret, H., Rajagopalan, R., Lepers, C., Gasciolli, V., Mallory, A.C., Hilbert, J., Bartel, D.P. & Crété, P. (2004). «Endogenous trans-acting siRNAs regulate the accumulation of Arabidopsis mRNAs». Molecular Cell 16 (1):
7.2 El Papel Del RNA En La Sintesis De Proteinas...!
El
hecho de que el ARN está involucrado en la síntesis de proteínas, se debe a las
investigaciones de Torbjörn Casperson y Jean Branchet a finales de los años
30s.Casperson confirmó, utilizando técnicas microscópicas, que en los
eucariontes el ADN está casi exclusivamente contenido en núcleo, mientras que
el ARN está distribuido en citoplasma. Branchet, quien trabajaba en el
fraccionamiento de organelos, llegó a conclusiones similares, basándose en
análisis químicos directos; encontró también que las partículas que contenían
ARN, eran ricas en proteínas; posteriormente, a estas partículas, se les
denominó ribosomas (ribo: de ribosa,
soma: de cuerpo). Ambos investigadores notaron que la concentración de estas
partículas estaba relacionada con la velocidad de síntesis de proteínas en la
célula.
Las
proteínas que han de construirse, están especificadas en el mRNA y se
sintetizan en los ribosomas. Esta idea surge del estudio de la inducción
enzimática, que es un fenómeno en el cual las bacterias varían sus velocidades
de síntesis de proteínas en respuesta a cambios en el ambiente. Este proceso
ocurre como consecuencia de la regulación de la síntesis de mRNA por proteínas
que se unen específicamente a los templados para el mRNA en el ADN.
La
comprencion de la sintesis de proteinas, el mas complejo de los mecansimos
biosinteticos. a sido uno de los mayores retos en al historia de la
bioquimica.Las proteínas que han de construirse, están especificadas en el mRNA
y se sintetizan en los ribosomas. Esta idea surge del estudio de la inducción
enzimática, que es un fenómeno en el cual las bacterias varían sus velocidades
de síntesis de proteínas en respuesta a cambios en el ambiente. Este proceso
ocurre como consecuencia de la regulación de la síntesis de mRNA por proteínas
que se unen específicamente a los templados para el mRNA en el ADN.
7.1 El Codigo Genetico...!
Tanto
el ARN como el ADN están compuestos por la combinación de cuatro bases
diferentes (se puede decir que es como un alfabeto de cuatro letras). ¿Cómo es
esta combinación? Si cada aminoácido estuviera codificado sólo por dos bases
habría un total de 42=16 posibilidades, pero esto no puede ser así, ya que los
aminoácidos encontrados en las proteínas son 20. Necesitamos combinar más
bases. Entonces, si combinamos tres bases (tripletes) para formar un
aminoácido, obtenemos un total de 64 combinaciones (43=64)… pero ahora “sobran”
44 tripletes. En esa situación se encontraron Har Gobind Khorana y Marshall
Nirenberg en la década de 1960.
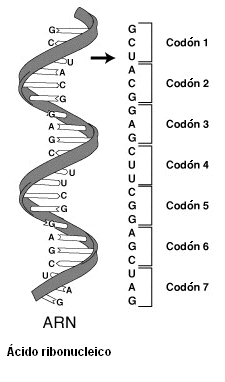 Este
código es casi universal: es el mismo en todos los organismos. Sin embargo, el
código genético mitocondrial es diferente del nuclear y se transmite de manera
independiente. De esto hablaremos en el siguiente título.
Este
código es casi universal: es el mismo en todos los organismos. Sin embargo, el
código genético mitocondrial es diferente del nuclear y se transmite de manera
independiente. De esto hablaremos en el siguiente título.
La
secuencia de aminoacidos de una proteina
en particular se constituye a partir de la transcripcion de la informacion
codificada en el mARN. este proceso esta acargo de las ribosomas.
Los
aminoacidos estan especialisados por los
codones del mARN consiste en los tripletes. la traduccion requiere moleculas
adaptadoras. Los tARN que reconoce los codones he inserta los aminoacidos en sus posiciones
secuenciales apropiadas en el polipeptido.
El
codigo genetico es degenerador tien multiples palabras de codigo para casi
todos los aminoacidos.
La
palabra estandar del codigo genético son universales en todas las especies, con algunos cambios
menores en las mitocondrias y en algunos organismos unicelulares.
LITERATURA CITADA:
Sintesis de poreinas. Candelas Manzano y Mª José Martínez
SEP
SNEST DGEST
INSTITUTO TECNOLÓGICO DE CIUDAD ALTAMIRANO
UNIDAD 7 TRADUCCION DEL RNA MENSAJERO.
Nombre del Alumno: GONZALEZ AGUIRRE CARLOS A.
Número de Control: 09930040
Carrera: Lic. Biología
Ciudad Altamirano, Gro. México. 04 de Mayo del 2012.
INTRODUCCION.
El ARN mensajero es el que
lleva la información para la síntesis de proteínas, es decir, determina el
orden en que se unirán los aminoácidos.
La síntesis de proteínas o
traducción tiene lugar en los ribosomas del citoplasma. Los aminoácidos son
transportados por el ARN de transferencia, específico para cada uno de ellos, y
son llevados hasta el ARN mensajero, dónde se aparean el codón de éste y el
anticodón del ARN de transferencia, por complementariedad de bases, y de ésta
forma se sitúan en la posición que les corresponde.
Una vez finalizada la síntesis
de una proteína, el ARN mensajero queda libre y puede ser leído de nuevo. De
hecho, es muy frecuente que antes de que finalice una proteína ya está
comenzando otra, con lo cual, una misma molécula de ARN mensajero, está siendo
utilizada por varios ribosomas simultáneamente.
Esta información está
codificada en forma de tripletes, cada tres bases constituyen un codón que
determina un aminoácido. Las reglas de correspondencia entre codones y
aminoácidos constituyen el código genético .
OBJETIVO.
Conocer
los eventos de síntesis proteica, la especificidad de la maquinaria enzimática
y su implicación en la farmacología (inhibición de la síntesis proteica por
antibióticos.
jueves, 10 de mayo de 2012
Conclusion Unidad 6
En esta unidad 6 logramos
entender lo que era el proceso de la transcripción genética. Puesto que en este
proceso explica como se trasforma el DNA en RNAm ademas esta metodo es muy
diferente entre las procariontes y eucariontes. También comprendimos los
procesos de maduración en el ARNm, ya que estas son muy interesantes, pues para
que esta molécula salga del núcleo debe de sufrir varias modificaciones como,
la adición de una caperuza en el extremo 5´
y una cola de muchas Adeninas conocida como cola Poly A. Estos procesos de maduración sirven para que
cuando salga el ARNm la vida media de este perdure más de lo
común, pues muchas enzimas que existen
en le citoplasma pueden degradarla fácilmente. Y por ultimo la molécula del
ARNm sufre unos cortes de intrones y empalme
de los exones conocido como Splicing, ya que, como todos sabemos, los
intrones nunca codifican proteínas por eso son cortados antes de salir del
núcleo.
Tarea...! Splicing..!
Es un proceso de corte y
empalme de ARN. Este proceso es muy común en eucariotas, pudiéndose dar en
cualquier tipo de ARN aunque es más común en el ARNm. El empalme alternativo de
transcriptos de ARN idénticos en diferentes tipos de células puede producir
diferentes moléculas de ARNm maduro que se traducen en diferentes polipéptidos.
La información genética
codificada en el AND se transcribe a una copia de ARN (transcripto primario).
Esta copia luego se modifica con la adición del casquete 5’ (CAP) y la cola de
poli-A, la escisión de los intrones y la unión de los exones (Splicing). El
mRNA maduro luego va al citoplasma, donde se traduce a proteínas.
Mecanismo molecular de
Splicing
Se trata de un mecanismo muy
exacto, pues de no serlo produciría un corrimiento del marco de lectura en el
mensaje transcripto. Los intrones son cortados del ARNm inmaduro por un sistema
específico que reconocen secuencias cortas dentro de él y que se encuentra
cerca de los límites con exones. Estas secuencias son llamadas "sitio
dador" (común en casi en todos los intrones), en el extremo 5’y "sitio
aceptor", en el extremo 3’.
El trabajo del corte y
empalme esta catalizado por una estructura pequeña, compuesta por
ribonucleoproteinas nucleares llamadas snRNPs, constituidas por pequeños
ARN nucleares asociado a proteínas. Su nombre es spliceosoma. Esta
estructura tiene a su cargo el reconocimiento de las secuencias mencionadas
anteriormente en los intrones y su posterior fijación. Luego se desarrollan una
secuencia de pasos que determinan el clivaje y ligado de los intrones y exones.
- El extremo 5’del intrón es clivado y unido a otros sitio interno del intrón, cercano a su extremo 3’ llamado"sitio de ramificación"
- Se produce el corte en el extremo 3’ del intrón y son empalmados los dos exones de cada lado, liberándose el ARNm maduro del spliceosoma.
- El intrón eliminado queda formando una estructura con forma de lazo, llamada "lariat", que posteriormente es degradado en el núcleo.
Se ha observado que ARNm
inmaduros idénticos del mismo gen se procesan en más de una forma. Esto
significa que existen diversos empalmes alternativos, los cuales desarrollaran
diversos ARNm maduros y por lo tanto distintos polipéptidos funcionales.
 Splicing alternativo: es un proceso de edición post-transcripcional
que se produce tras la obtención del ARN mensajero primario. El Splicingalternativo
permite que en un mismo gen pueda estar codificada la información necesaria
para sintetizar distintas proteínas ya que mediante este proceso a partir de un
mismo mensajero primario pueden obtenerse varias secuencias de ARN mensajero
maduro dependiendo de cuáles sean los exones que se combinen. El mecanismo deSplicing alternativo
es una de las maneras de originar distintas isoformas funcionales de una misma
proteína en diferentes tejidos o compartimentos celulares.
Splicing alternativo: es un proceso de edición post-transcripcional
que se produce tras la obtención del ARN mensajero primario. El Splicingalternativo
permite que en un mismo gen pueda estar codificada la información necesaria
para sintetizar distintas proteínas ya que mediante este proceso a partir de un
mismo mensajero primario pueden obtenerse varias secuencias de ARN mensajero
maduro dependiendo de cuáles sean los exones que se combinen. El mecanismo deSplicing alternativo
es una de las maneras de originar distintas isoformas funcionales de una misma
proteína en diferentes tejidos o compartimentos celulares.
El Splicing alternativo
añade complejidad a los mecanismos de regulación de la expresión génica ya que
permite codificar mayor número de proteínas con el mismo número de genes.
Mediante estudios realizados con métodos informáticos se estima que en humanos
cerca de un 50% de los productos de los genes son susceptibles de ser
procesados por Splicing alternativo.
Splicing autocatalitico Los científicos
estadounidenses Thomas Cech y Sidney Altman descubrieron que, además del Splicing que
ocurre normalmente en el núcleo de células eucariontes y produce mRNA
maduro, otro grupo de RNA sufre un tipo de Splicing todavía
más espectacular. Este tipo de mecanismo, la autocatálisis del RNA, fue
observado por primera vez por Cech y su grupo cuando estudiaban al protista
unicelular Tetrahymena y uno de sus RNA ribosómicos. Los
científicos pudieron demostrar que un intrón tiene una actividad
catalítica de tipo enzimático, que lleva a cabo la escisión y el empalme.
Aunque los RNA autocatalíticos no son comunes, luego se fueron encontrando
otros ejemplos de este tipo de mecanismo de Splicing en varios
organismos, en general en RNA codificados por genes mitocondriales (no en
animales vertebrados) o de cloroplastos, en algunos genes nucleares de células
eucariontes como protistas y en algunos genes de bacteriófagos. El
descubrimiento de que el RNA puede actuar como catalizador hace más
fácil el imaginar cómo comenzó la vida. Según Bruce M. Alberts "uno
sospecha que un primer acontecimiento crucial fue la aparición de una molécula de
RNA que podía catalizar su propia replicación".
LITERATURA CITADA:
http://www.botanica.cnba.uba.ar/Pakete/Dibulgeneral/Splicing/Splicing.htm
http://www.curtisbiologia.com/B1983
6.2.2 Modificaciones postranscripcionales del RNA mensajero
Los productos inmediatos de
la transcripción se denominan transcritos primarios y no son necesariamente
funcionales; muchos de ellos son específicamente alterados en varias formas.
Estas modificaciones no son iguales para todos los tipos de ARN, cada uno tienen
sus propias particularidades.Los procesos de maduración son los que llevan a
los transcritos primarios a convertirse en transcritos maduras.
Los procesos de maduración
son los que llevan a los transcritos primarios a convertirse en transcritos
maduras.
Todos estos procesos ocurren
mientras el RNA se está transcribiendo, demostrándose en algunos casos la
interdependencia entre transcripción y maduración.
Las diferencias entre los
procariotas y las eucariotas relativas a la maduración del RNA se centran en el
mRNA, además del ayuste, sufre una serie de modificaciones en 5’ y en 3’ que,
de forma sucesiva o simultánea, van a ocurrir en el núcleo.
Splicing:
Todos los Pre- ARN m sufren un proceso denominado Splicing, que tiene como
finalidad eliminar a los Intrones (secuencias no codificantes). Este proceso
ocurre en el núcleo y da origen al ARN m que será desplazado al citoplasma,
para ser partícipe del proceso de Traducción. En el Splicing son descartados
del 50% al 90% del Pre-ARNm (transcripto primario). Los elementos remanentes
(exones) son unidos o ensamblados. El número de intrones, generalmente
sobrepasa al de los Exones.
Cola Poly A:
En el extremo 3’, según se mencionó antes, los ARNm Eucariotas contienen una
secuencia de 20 a 200 nucleótidos que poseen como base nitrogenada a la
Adenina. La secuencia Poly A no está codificada en el ADN, sino que es añadida
al ARN después de finalizada la transcripción. La función de la Cola Poly A es
la de aumentar la estabilidad de los ARNm.
 Los principales mecanismos de
regulación de la expresión génica, recaen en el proceso de transcripción. Es
así como, la célula podrá “elegir” qué genes transcribe y cuáles no,
dependiendo de las necesidades metabólicas reinantes en ese momento. Cada día se
postulan más mecanismos por los cuales la transcripción puede ser regulada,
nosotros nos dedicaremos al estudio del mecanismo clásico de regulación: el
Modelo del Operón.
Los principales mecanismos de
regulación de la expresión génica, recaen en el proceso de transcripción. Es
así como, la célula podrá “elegir” qué genes transcribe y cuáles no,
dependiendo de las necesidades metabólicas reinantes en ese momento. Cada día se
postulan más mecanismos por los cuales la transcripción puede ser regulada,
nosotros nos dedicaremos al estudio del mecanismo clásico de regulación: el
Modelo del Operón.
El Modelo del Operón fue
descripto en células Procariotas, por los investigadores franceses Francois
Jacob y Jacques Monod, quienes se hicieron acreedores del Premio Nobel en el
año 1965.
Tal como postularan Jacob y
Monod, los grupos de genes que codifican para proteínas asociadas se encuentran
en unidades conocidas como Operones. Un Operón está constituido por: un
Promotor, un Operador y Genes Estructurales. El Promotor, según vimos en el
apartado anterior, es el lugar del ADN en el cual se une la ARN polimerasa para
iniciar la transcripción. El operador es una secuencia de nucleótidos que se
encuentra interpuesto entre el promotor y los genes estructurales. Los Genes
Estructurales son las porciones de ADN que codifican para la síntesis de las
proteínas asociadas metabólicamente.
miércoles, 9 de mayo de 2012
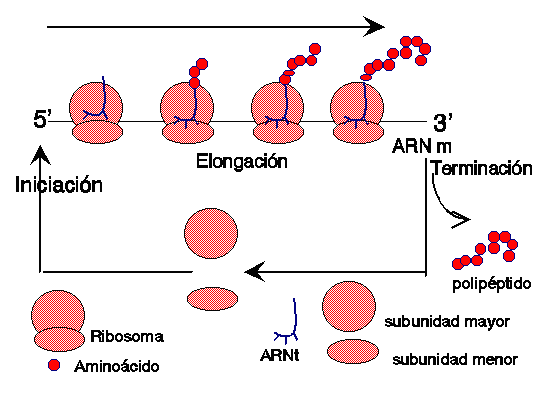
6.2.1 Etapas De Sintesis De Proteinas.
Dividiremos
el proceso de traducción en cuatro etapas:
1.
Activación de los aminoácidos
2.
Iniciación
3.
Elongación
4.
Terminación
1-Activación
de los aminoácidos
Para
que el aminoácido sea unido al correspondiente ARNt se necesitan dos reacciones
químicas
que demandarán energía. La primera reacción es la que aporta la energía
necesaria
para que la unión se produzca. Se escinde la molécula de ATP, se liberan dos
grupos
fosfatos y se forma un complejo entre el AMP y el aminoácido en cuestión. Este
complejo
Aminoácido- AMP – Enzima, se mantiene intacto hasta que se encuentra con
la
molécula de ARNt correspondiente. En la segunda reacción, la molécula de AMP se
desprende
de la enzima, formándose un enlace entre el aminoácido y la porción 3´ del
ARNt.
Gracias a estas reacciones, el aminoácido queda unido al ARNt y este complejo
queda
listo para formar parte de la síntesis proteica.
Aminoácido
+ ATP____________> Aminoacil-AMP
+ PPi
Aminoacil-AMP
+_____________ > ARNt Aminoacil-
ARNt + AMP
2-Iniciación:
Una
vez que los aminoácidos son unidos a su correspondiente ARNt, están listos para
comenzar
la síntesis de proteínas. La primera etapa, la iniciación, comienza cuando la
sub
unidad menor del ribosoma se une al ARNm, por su extremo 5´. Cada ARNm
posee,
en forma previa al primer codón, una secuencia de bases complementaria a una
secuencia
de consenso de la sub unidad menor del ARNr.
Una
vez sucedido este, el primer aminoacil-ARNt se une, por medio de su Anticodón,
al
primer
codón del ARNm. Se ha observado que, habitualmente, el primer codón del
ARNm
es el AUG. Si recurrimos al código genético veremos que, en consecuencia, el
primer
aminoácido incorporado a la cadena peptídica en formación es la Metionina. Sin
embargo
la Metionina, una vez finalizada la síntesis proteica, puede ser
removida.Complejo de Iniciación de la traducción. Para que se veaFactores de
Iniciación. En Procariotas se conocen tres.
La
combinación entre el ARNm, la sub unidad ribosómica menor y el aminoacil-ARNt
es
conocida como el posibilitada la formación del Complejo de Iniciación, es
necesaria la participación de ciertas proteínas denominadas de estos factores,
mientras que en Eucariotas el número conocido hasta el momento
3-
Elongación
Al
iniciarse esta etapa en el segundo codón del ARNm. Un aminoacil – ARNt que
posea su Anticodón complementario al segundo codón del ARNm, ingresará al vez
que ambos sitios de la sub unidad mayor del ribosoma se encuentran cubiertos,
la enzima de ésta sub unidad, la Peptidil Transferasa, realizará el primer
enlace peptídico entre los aminoácidos portados por ambos ARNt. En dicho enlace
interviene el grupo Sitio A, de la sub unidad mayor del ribosoma, se
encuentrasitio A del ribosoma. Una Amino anterior.
Así
cada vez que ingresa un ARNt cargado al grupo COOH del aminoácido de la cadena
en crecimiento y el grupo NH Aminoácido del primer ARNt, que estaba ocupando el
adherido
al primer aminoácido y es liberado al medio. El ribosoma se desplaza un
codón
hacia el extremo 3´ del ARNm. En consecuencia, el segundo ARNt incorporado
en
el hasta el momento. De esta manera, en el ingresando al mismo un tercer
aminoacil - ARNt que posee un Anticodón complementario al mencionado triplete
de bases del ARNm.
Nuevamente,
la enzima ribosómica realiza la unión peptídica entre el segundo y tercer
aminoácido
incorporado. El segundo ARNt es liberado al medio, quedando el tercer
ARNt
con los tres aminoácidos unidos a él. De la misma manera que fue realizado en
la
etapa
anterior, el ribosoma se desplaza un codón hacia el extremo 3´ del ARNm,
quedando
ahora el tercer ARNt en la posición P de la sub unidad mayor. El ribosoma
seguirá
corriéndose hacia el extremo 3´ del ARNm y los aminoácidos serán
incorporados
de igual forma a lo largo de toda esta etapa.
Una
de las formas por las cuales se logra que la traducción se lleve a cabo a una
velocidad
mayor, es mediante el accionar de varios ribosomas a lo largo del ARNm. La
estructura
que forma esta multitud de ribosomas, traduciendo un mismo ARNt, es
conocida
como:
4-
Terminación
El
proceso descripto se llevará a cabo, en forma repetida, hasta que en la
molécula de
ARNm
se encuentre un codón sin sentido. Tal cual lo expresamos con anterioridad, se
conocen
tres codones sin sentido, el UAG, el UUA y el UGA. No existe ningún ARNt
que
sea complementario a estos codones de ARNt cadena polipeptídica se desprenderá
y las dos sub unidades ribosómicas se separarán.
Finaliza
así el proceso de traducción, obteniéndose como producto la cadena
polipeptídica
en forma libre. Esta cadena proteica se libera así al citoplasma, pudiendo
luego
sufrir procesos post-traduccionales que la modifiquen activándola o desactivándola.
VIDEO SOBRE LA TRANSCRIPCION EN EUCARIOTAS.
LITERATURA CITADA:
Sintesis de Proteinas PDF Candelas Manzano y Mª José Martínez 13 .
Suscribirse a:
Entradas (Atom)


















.gif)

